Deep Learning und Digitale Pathologie
Die digitale Pathologie hat sich in letzter Zeit aufgrund von Fortschritten in der Technologie und bei den Vorschriften weithin durchgesetzt. Im Wesentlichen erzeugt ein Scanner in der digitalen Pathologie eine digitale Kopie des traditionellen Glasobjektträgers, die auf einem lokalen oder cloud basierten Server gespeichert und überall mit einem Computer und einer Internetverbindung bearbeitet werden kann. Ihr Einsatz für die Primärdiagnose hat die Pathologie revolutioniert und prägt die Zukunft dieser Disziplin.
Das DigiPath-Team beschäftigt sich mit verschiedenen Herausforderungen im Bereich der digitalen Pathologie. Dazu gehört z. B. die Klassifizierung von Tumor- und Nicht-Tumorgewebe oder von verschiedenen Tumortypen auf Basis digitaler Mikroskopieaufnahmen. Diese Aufgabe würde normalerweise eine umfangreiche mikroskopische Auswertung durch Patholog*innen erfordern. In unserem Team entwickeln wir dafür leistungsfähige und robuste Methoden, die sowohl Deep Learning als auch klassische maschinelle Lernverfahren nutzen.
Forschungsgebiete
- Semantische Segmentierung
- Interpretierbarkeit/Erklärbarkeit von Unet-ähnlichen neuronalen Netzen
- Stain-Normalisierung mit GANs
- Normalizing-Flows und invertierbare UNet für die Erzeugung synthetischer Daten
- Training mit schwachen Etiketten + Differentialdiagnose
Leitung
DigiPath-Viewer
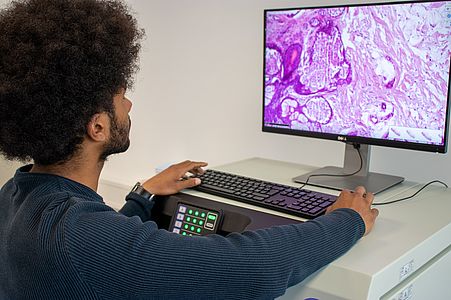
Das Digipath-Team entwickelt außerdem einen "DigiPath Viewer", eine Web-Anwendung, mit der digitale Mikroskopiebilder annotiert und die Ergebnisse der Modelle angezeigt werden können. Der "DigiPath Viewer" ermöglicht die Visualisierung der Deep Learning Ergebnisse und wird die Arbeit der Patholog*innen vereinfachen. Einige der Hauptmerkmale sind:
- Integration mit Stylus-Stift für natürliche und präzise Annotationen.
- Ein halbautomatisches Annotationswerkzeug, das den Annotationsprozess beschleunigt. Das Annotationspolygon kann durch Ziehen/Löschen von Punkten einfach angepasst werden.
- Visualisierung von automatischen Tumorvorhersagen der künstlichen Intelligenz als Overlay auf den Originalbildern. Die Transparenz des Overlays sowie der Schwellenwert zur Unterscheidung zwischen Tumor- und Normalgewebe können einfach angepasst werden.
Teilnahme an Challenges
Unser Team belegte den 9. Platz in der inoffiziellen Rangliste der CAMELYON 17 Challenge, die auch nach den offiziellen Einreichungsfristen offen blieb und mehr als 100 Einreichungen von den besten Unternehmen und Forschungsgruppen im Bereich der digitalen Pathologie erhielt. Das Ziel dieser Challenge ist die Entwicklung einer Methode zur automatischen Erkennung und Klassifizierung von Brustkrebsmetastasen in digitalen Mikroskopiebildern von histologischen Lymphknotenschnitten.
Team
Projekte
Veröffentlichungen
J. Le Clerc Arrastia, N. Heilenkötter, D. Otero Baguer, L. Hauberg-Lotte, T. Boskamp, S. Hetzer, N. Duschner , J. Schaller , P. Maaß.
Deeply Supervised UNet for Semantic Segmentation to Assist Dermatopathological Assessment of Basal Cell Carcinoma.
MDPI Journal of Imaging, 71 7(4), Meisenbach Verlag, Bamberg, 2021. DOI: https://doi.org/10.3390/jimaging7040071
J. Behrmann, C. Etmann, T. Boskamp, R. Casadonte, J. Kriegsmann, P. Maaß.
Deep Learning for Tumor Classification in Imaging Mass Spectrometry.
Bioinformatics, 34(7):1215-1223, Oxford University Press, 2018. DOI: 10.1093/bioinformatics/btx724
C. Etmann, M. Schmidt, J. Behrmann, T. Boskamp, L. Hauberg-Lotte, A. Peter, R. Casadonte, J. Kriegsmann, P. Maaß.
Deep Relevance Regularization: Interpretable and Robust Tumor Typing of Imaging Mass Spectrometry Data.
Zur Veröffentlichung eingereicht, online unter: https://arxiv.org/abs/1912.05459